Cohortes en santé : données ouvertes en imagerie médicale
Résumé de section
-
Ce cours examine en profondeur les cohortes en santé et l'utilisation des données d'imagerie médicale en recherche. Il couvre les enjeux techniques, juridiques et éthiques de l'ouverture des données d'imagerie au sein de cohortes, ainsi que les défis liés à l'évolution des librairies de programmation et de l'environnement d'exécution dans le contexte des pipelines de traitement.
Objectifs
- Comprendre la structure du format DICOM.
- Identifier les obligations et contraintes en termes de RGPD.
- Identifier les étapes des pipelines de traitement.
- Identifier les techniques pour interagir avec les données.
Ce cours est en libre accès !
Aucune création de compte ou d'inscription n'est nécessaire, toutefois vous ne pourrez le parcourir qu'en lecture seule.
Pour participer aux activités (exercices, forum...), vous devez vous inscrire au cours
S'inscrire au cours -
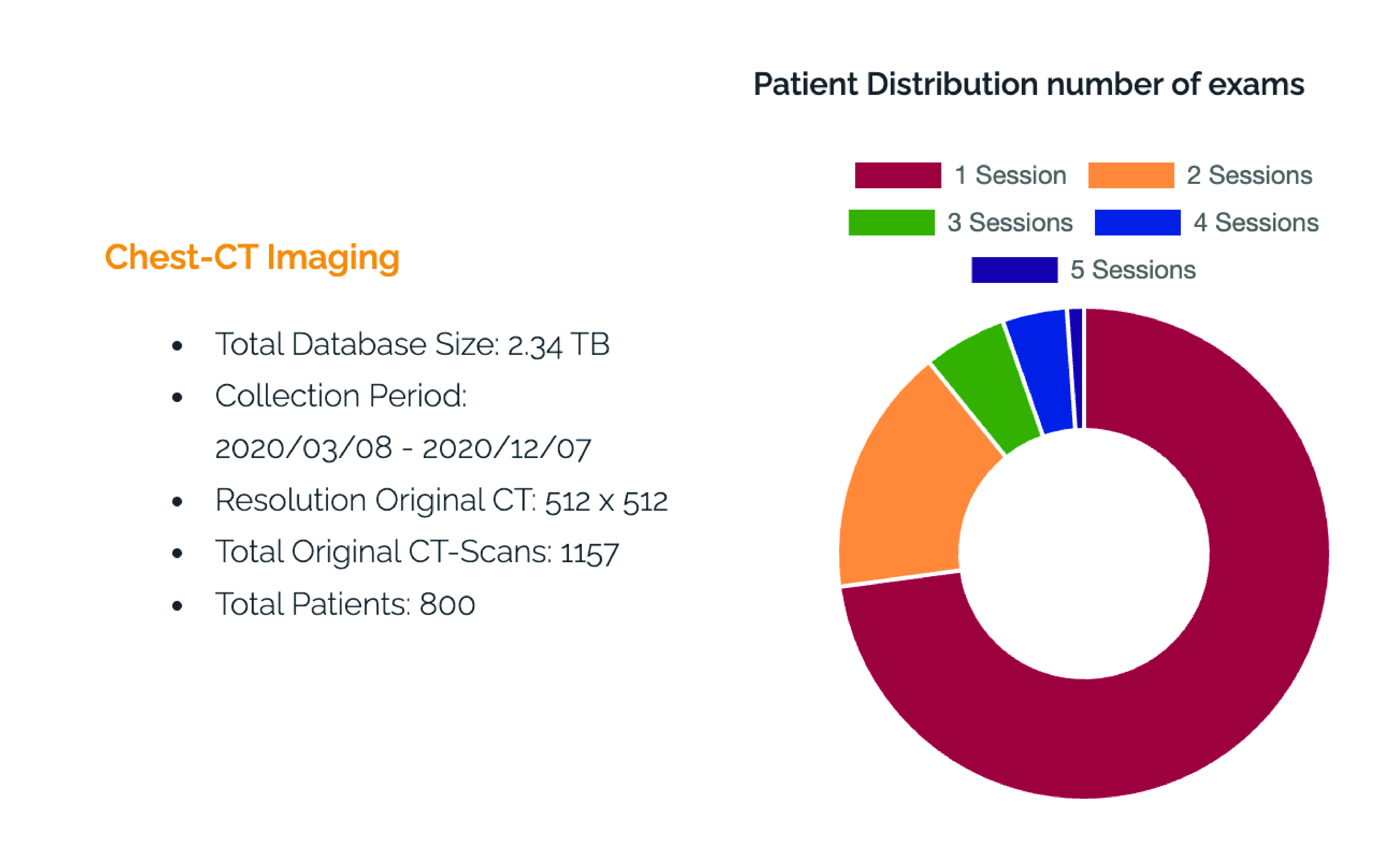
Qu'est-ce qu'une cohorte et quelles sont les particularités de ces données ?
-
DICOM est un format standard qui a la particularité de contenir données et métadonnées au sein d'un même fichier.
-
Comment anonymiser des images médicales sans perdre d'informations utiles ?
-
Que dit la loi concernant le traitement des données d'imageries médicales ?
-
Cet exercice vous permet de tester l'anonymisation d'un fichier DICOM à l'aide de la librairie PyDicom.
-
Les pipelines de traitement sont des étapes nécessaires pour exploiter les images médicales.
-
Il existe de nombreux challenges en imagerie médicale qui sont souvent organisés lors de compétitions, de congrès ou de conférences. Ces challenges servent de plateformes pour évaluer et comparer les méthodes de traitement d'images médicales.
-
Sept questions revenant sur des notions clés abordées dans ce cours.
-
-
Ce forum est disponible pour lancer des discussions entre vous ou poser des questions relatives au cours.
-
-
Autres suggestions